Publicada na Revista Nature em 8 de outubro, a estrutura metodológica MetaGraph promete transformar a forma como cientistas pesquisam e analisam sequências de DNA, RNA e proteínas.
Inspirado no funcionamento de buscadores como o Google, o MetaGraph permite explorar informações genéticas por palavras-chave, facilitando o trabalho de pesquisadores em diversas áreas das ciências da vida.
Entenda como essa ferramenta funciona e por que ela pode transformar o futuro das pesquisas biológicas.
O que é o MetaGraph?
O MetaGraph tem uma linha de pesquisa similar ao Google, ao permitir o acesso rápido a informações por meio de palavras-chave. Além disso, a metodologia de funcionamento do repositório permite uma pesquisa mais aprofundada, já que podemos consultar uma sequência e ter acesso, também, a variáveis e conjuntos aproximados da amostra.
Essa relação permite uso de diferentes representações de gráficos e anotações de forma intercambiável, adaptando-se a diferentes requisitos de armazenamento e tarefas de análise e permitindo a fácil adoção de novos desenvolvimentos algorítmicos.
Falando em termos científicos, o MetaGraph é uma importante ferramenta que permite que os pesquisadores acessem dados genômicos de forma rápida (com ferramentas de busca de última geração), diversificada (uma vez que conta com códigos de diversas espécies animais, vegetais, fúngicas e virais), eficientes (já que o repositório usa sistemas de limpeza e alinhamento mostrando que os gráficos de amostra individuais representam com precisão seus respectivos conjuntos de leitura de entrada) e barata (o espaço ocupado pelos índices MetaGraph é 3×10-15 menor do que o das outras abordagens avaliadas, o que diminui substancialmente o custo de armazenamento).
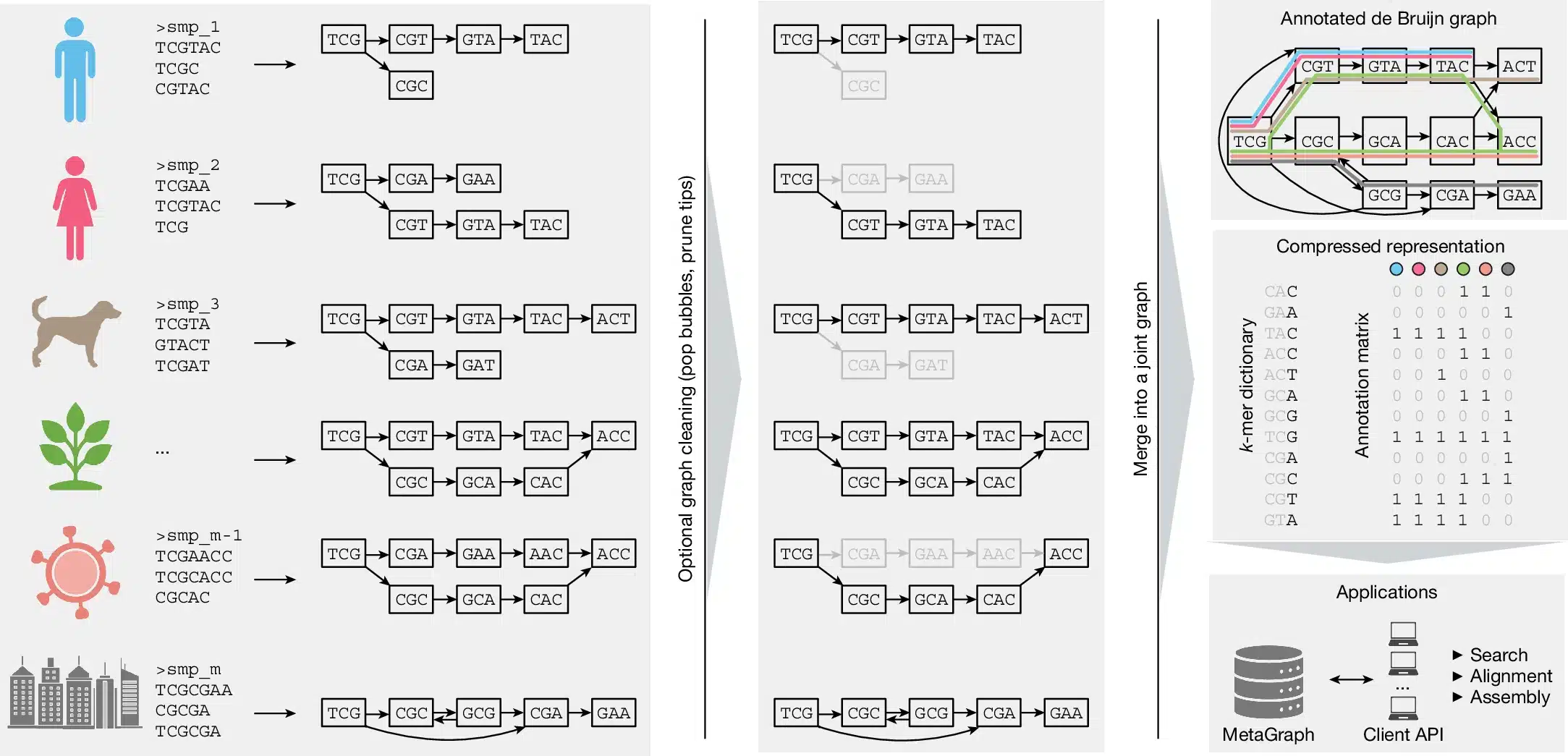
Qual o método usado por ele?
A indexação é aplicada a conjuntos de leitura que se originam do sequenciamento de amostras biológicas, onde cada rótulo de anotação codifica um ID de amostra. Para uma amostragem correta, são cumpridas 3 etapas:
- Pré-processamento de dados (gráficos de Bruijn separados (gráficos de amostra) de amostras brutas de entrada)
- Construção de gráficos (todos os gráficos de amostra obtidos no primeiro estágio são mesclados em um único gráfico de Bruijn)
- Construção da informação (construímos as colunas da matriz de anotação para indicar a associação de diferentes k-mers em seus respectivos gráficos de amostra)
Aqui você pode acessar todos os softwares e databases usados!
Por que ele foi criado?
Curiosamente, a quantidade de dados que existe dificulta a pesquisa científica, já que as leituras de sequenciamento bruto são fragmentadas, barulhentas e numerosas demais para serem pesquisadas diretamente. Para isso, a ferramenta de pesquisa facilita o acesso e diminui a probabilidade de erro dos cientistas.
Os gráficos matemáticos usados são cruzados e sobrepostos para que informações não sejam duplicadas ou incluídas de forma contrária. Os desenvolvedores do MetaGraph queriam desenvolver um mecanismo de busca para essas sequências, no qual os usuários usam prompts de texto para pesquisar esses arquivos integrados de dados brutos.
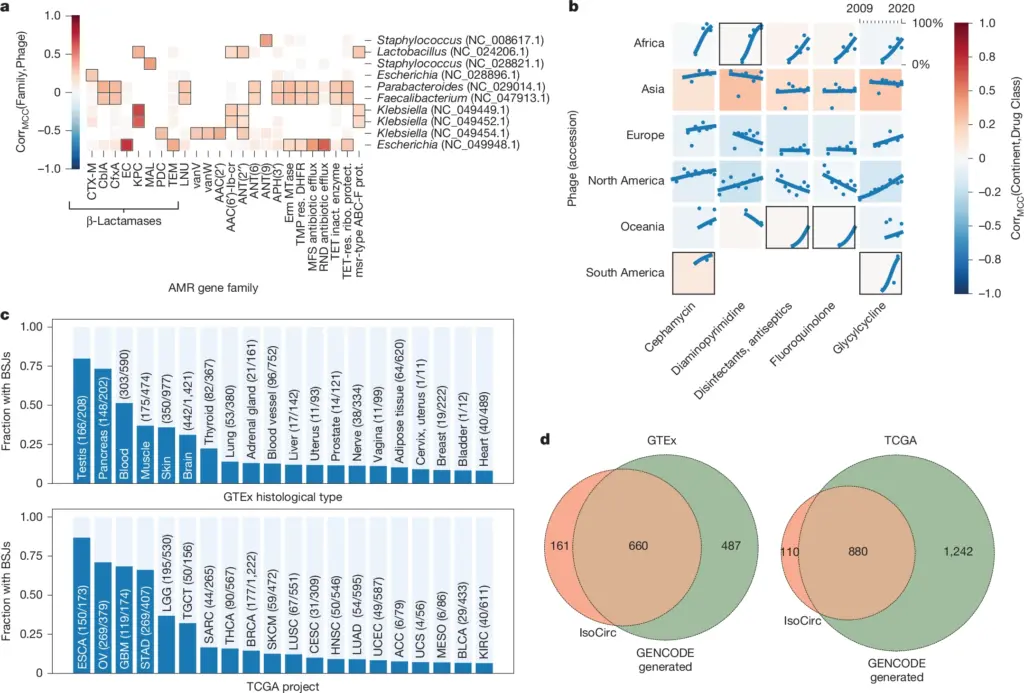
Quem pode usar?
O MetaGraph foi desenvolvido para ser acessível a pesquisadores em ciências da vida e outras comunidades para exploração e pesquisa de dados de sequenciamento de forma fácil e econômica. Além disso, o MetaGraph Online oferece uma interface web intuitiva, permitindo que qualquer usuário cole uma sequência e a pesquise em um índice selecionado. Isso sugere que tanto especialistas quanto não especialistas podem utilizar a ferramenta para diferentes finalidades.
Quais os benefícios?
Primariamente, os dados foram pensados para suprir as pesquisas sobre resistência a antibióticos, contudo o potencial do repositório pode ser bem mais amplo. Algumas possíveis aplicações do sistema são:
- Pesquisa do Microbioma: Estudo da composição e função de comunidades microbianas em vários ambientes
- Monitoramento Ambiental: Rastreamento da presença de organismos ou poluentes específicos em amostras ambientais
- Descoberta e vigilância viral: Dada a sua capacidade de procurar rapidamente sequências específicas de DNA/RNA, ele pode ser usado para identificar novos vírus ou rastrear a propagação de vírus conhecidos.
- Medicina Personalizada: Análise do microbioma ou genoma de um paciente para identificar potenciais alvos de medicamentos ou prever sua resposta ao tratamento.
O método que te aprova!
Quer alcançar a aprovação nas provas de residência médica? Então seja um MedCofer! Aqui te ajudaremos na busca da aprovação com conteúdos de qualidade e uma metodologia que já aprovou mais de 20 mil residentes no país! Por fim, acesse o nosso canal do youtube para ver o nosso material.